Machine moléculaires
En combinant génétique moléculaire, microscopie et analyses bioinformatiques, des chercheurs de Chimie Bactérienne du CNRS, en collaboration avec la plateforme de Biologie Intégrative et Computationnelle (BIC) du BIAM, ont mis en lumière un système d’attaque bactérienne d’une étonnante sophistication : une bactérie prédatrice est capable de moduler l’extrémité de ses pili (petits filaments à la surface de la cellule) pour mieux reconnaître et s’adapter à ses proies. Cette découverte ouvre des perspectives nouvelles en microbiologie, en infectiologie et en biotechnologie.
Dans le cadre de ses recherches sur les bases moléculaires, cellulaires et évolutives de la prédation bactérienne en utilisant Myxococcus xanthus comme organisme modèle, des scientifiques de l’Institut microbiologie, bioénergies et biotechnologie (IM2B) dirigés par les Dr. Julien Herrou et Tâm Mignot (CNRS) viennent de publier des recherches qui élargissent notre compréhension des interactions microbiennes.
Une stratégie de prédation unique qui adapte l’arme à la cible
Certaines bactéries, comme Myxococcus xanthus, pratiquent la prédation bactérienne en attaquant directement d’autres bactéries pour s’en nourrir. Ce mode de vie original repose sur un contact direct entre le prédateur et sa proie. L’article paru dans le journal Nature communications ce mois-ci présente des avancées majeures dans la compréhension de ce processus.
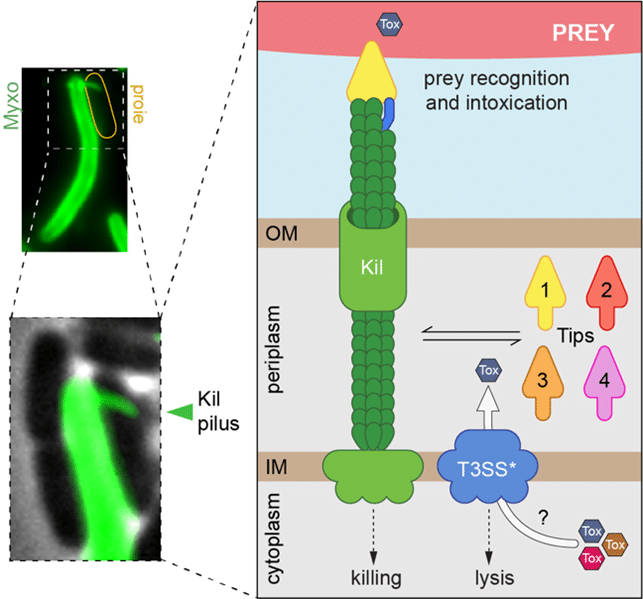
L’étude menée par le Laboratoire de Chimie Bactérienne (LCB) situé sur le campus Joseph Aiguier de Marseille, met en lumière certains mécanismes moléculaires associés à la reconnaissance et la prédation de bactéries bien plus sophistiqués qu’on ne le pensait. Les chercheurs se sont intéressés à un type particulier de pili bactériens—de fins filaments situés à la surface de la cellule—appelés pili Tad. Plus précisément, ils ont étudié une version impliquée dans la prédation, baptisée pili Kil. Ces filaments sont produits localement, exactement au point de contact entre la bactérie et sa proie. Ils sont constitués d’assemblages de nombreuses sous-unités protéiques appelées pilines.
Mais la découverte majeure de cette étude a dévoilé que l’extrémité du pilus était capable de changer de composition : quatre complexes différents de « pilines mineures » peuvent en effet s’associer pour former un embout, ou tip, modulaire. Ces embouts interchangeables permettent à la bactérie d’adapter finement son pilus en fonction de la proie rencontrée, tel un couteau suisse dont l’outil change selon la tâche à effectuer.
Fait intéressant, le bon fonctionnement de ces embouts dépend aussi d’un autre système intracellulaire appelé T3SS*(Type III Secretion System « sans aiguille »). Ce système est connu pour permettre à certaines bactéries pathogènes d’injecter des protéines dans des cellules hôtes, mais ici, il semble coopérer avec les pili Kil pour permettre la prédation.
Une ubiquité et une diversité révélée par la bioinformatique
C’est grâce l’expertise de la plateforme BIC du BIAM que cette stratégie s’est révélée bien plus répandue dans le monde bactérien qu’on ne le pensait, y compris chez certains pathogènes, « ce qui ouvre des perspectives pour comprendre d’autres mécanismes d’interaction cellule-cellule chez les microbes » pointent Marine Bergot et Caroline Monteil, bioinformaticiennes et scientifiques au sein de la plateforme BIC du CEA/BIAM. « L’analyse de plusieurs milliers de génomes complets a montré que des structures similaires aux pili Kil avec des embouts modulables existent. La diversité de ces embouts est cependant très loin d’être quantifiée. Ces pili adaptables à façon observés chez Myxococcus xanthus pourraient jouer un rôle important dans les interactions des pathogènes avec les cellules hôtes ou d’autres bactéries spécifiques ».
Si de tels pili étaient impliqués dans la virulence de certaines bactéries pathogènes, cela pourrait offrir de nouvelles cibles pour des traitements innovants. En bloquant ces pili ou leurs embouts modulaires, on pourrait désarmer les bactéries sans les tuer, limitant ainsi la pression sélective qui favorise les résistances. Autre alternative, cette capacité de ciblage pourrait être exploitée pour délivrer précisément des agents antimicrobiens à certaines bactéries — un peu comme un antibiotique guidé.
Aujourd’hui, le Dr. Julien Herrou poursuit la caractérisation fonctionnelle des embouts modulables appelés pilines mineures dans plusieurs bactéries modèles chez lesquelles ce système a également été identifié : « Il reste beaucoup à faire à la suite de cette découverte fascinante » souligne-t-il.
Cette avancée illustre la force des approches intégrées mêlant biologie moléculaire, imagerie avancée et bioinformatique à grande échelle, pour révéler les stratégies adaptatives du monde microbien, et ouvre des pistes prometteuses en matière de virulence, de prédation bactérienne et de développement d’outils thérapeutiques ciblés.
Références
Julien Herrou, Laetitia My, Caroline Monteil, Marine Bergot, Rikesh Jain, et al.. Tad pili with adaptable tips mediate contact-dependent killing during bacterial predation. Nature Communications, 2025, 16 (1), pp.4425. ⟨10.1038/s41467-025-58967-0⟩. ⟨hal-05086363⟩