Communiqué de presse
La transcription joue un rôle clef au sein des cellules en assurant la copie et l’amplification de l’information portée par le matériel génétique (ADN), sous la forme d’ARN messagers (ARNm). Son étude est cruciale pour comprendre le fonctionnement des organismes vivants. En effet, que ce soit chez les animaux ou les végétaux, 5 à 6 % des gènes sont impliqués dans la régulation de ce phénomène, illustrant clairement son importance biologique.
Découverte en 1961, la transcription de l’ARNm était jusqu’à présent majoritairement observée sur cellules animales isolées et fixées, grâce à des techniques de pointe notamment développées depuis de nombreuses années par une équipe de l’Institut de génique humaine de Montpellier (CNRS/Université de Montpellier). L’absence de mouvement chez les plantes a enfin permis de passer à une nouvelle échelle de complexité sur l’ensemble des cellules d’un organisme vivant. En effet, une équipe de l’Institut de Biosciences et biotechnologies d’Aix-Marseille (BIAM CEA/CNRS/AMU)[1] est parvenue à observer et filmer en temps réel la transcription de l’ARNm dans l’ensemble des cellules d’une plante.
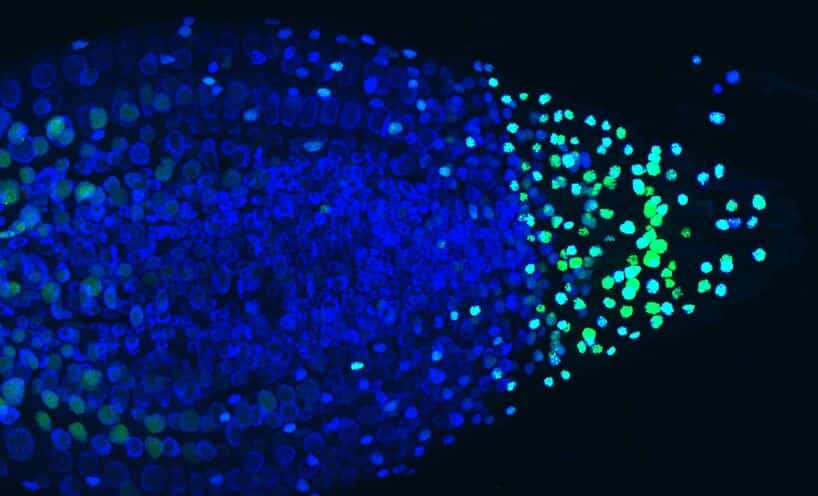
Ces travaux facilitent désormais le décryptage des mécanismes de nombreux phénomènes contrôlant le développement et l’adaptation des plantes en réponse aux contraintes de leur environnement.
Ainsi, l’utilisation des techniques conventionnelles montraient auparavant que les plantes répondaient en moins de 20 minutes aux apports de phosphate (un de leurs principaux nutriments). L’utilisation de ces nouvelles approches a démontré qu’il ne fallait en réalité que 3 à 5 minutes à la plante pour réagir aux variations de son environnement nutritif.
En outre, ces analyses ont également révélé une importante hétérogénéité de la transcription entre des cellules censées assurer des fonctions identiques : une réalité que les scientifiques étaient loin de soupçonner !
Comment cela fonctionne t-il ?
[1] Ces travaux ont également impliqué des scientifiques du Laboratoire génome et développement des plantes (CNRS/Université de Perpignan – Via Domitia), Dynamique cellulaire physiologique et pathologique (CNRS/Institut Pasteur), du Laboratoire de physiologie cellulaire et végétale (CNRS/CEA/INRAE/Université Grenoble Alpes) et l’Institut de génétique humaine à Montpellier (CNRS/Université de Montpellier).
Pour pouvoir visualiser des molécules d’ARNm (soit des objets de 10 à 500 nm), les scientifiques ont exploité l’expression d’une protéine virale fusionnée à un marqueur fluorescent. Cette protéine reconnaît une séquence spécifique d’ARN introduite dans le gène ciblé de la plante dans le gène que l’on souhaite étudier. Lorsque ce dernier est transcrit en ARN, il agrège ainsi 256 protéines fluorescentes sur l’ARNm produit. Ce système permet d’observer directement les zones de transcription de l’ADN au sein du noyau des cellules (Fig1).
Une prouesse technique
Les plantes se révèlent être un matériel expérimental de choix dans l’étude des cellules : à l’inverse des organismes animaux, ces dernières sont en effet facilement immobilisables.
Des technologies récentes comme la microfluidique autorisent le contrôle fin de la nutrition apportée aux plantes, tout en étant compatible avec des observations microscopiques. Pour ce faire, un polymère est coulé sur une lame de microscopie. Il intègre de fins canaux ménagés pour permettre la croissance et l’alimentation des racines tout en limitant au maximum leur mouvement.
L’équipe du BIAM a combiné cet outil avec de l’imagerie à haute résolution et haute vitesse, permettant l’acquisition de 200 images en quelques secondes. C’est ainsi qu’ils ont pu accéder à l’ensemble des couches cellulaires de la racine et suivre en temps réel les modifications survenant lorsqu’une plante est alimentée par du phosphate.
Pour visualiser la transcription à une résolution jamais atteinte :
https://imjoy.io/lite?plugin=muellerflorian/hani-ms2:hani-ms2-sample-3
https://imjoy.io/lite?plugin=muellerflorian/hani-ms2:hani-ms2-sample-1
AFFILIATIONS
- 1Aix Marseille Univ, CEA, CNRS, BIAM, UMR7265, SAVE (Signalisation pour l’Adaptation des Végétaux à leur Environnement), Saint-Paul lez Durance, France.
- 2Agroinnovation International-TIMAC AGRO, Groupe Roullier, Saint-Malo, France.
- 3Department of Animal, Plant and Soil Sciences, Australian Research Council Centre of Excellence in Plant Energy Biology, School of Life Sciences, La Trobe University, Bundoora, Victoria, Australia.
- 4UMR5096 CNRS/Université de Perpignan, Laboratoire Génome et Développement des Plantes, Perpignan, France.
- 5Unité Imagerie et Modélisation, Institut Pasteur and CNRS UMR 3691, Paris, France.
- 6MRI, BioCampus Montpellier, CRBM, Univ. Montpellier, CNRS, Montpellier, France.
- 7UMR 5168 CNRS-CEA-INRA-Université Grenoble Alpes, Laboratoire de Physiologie Cellulaire et Végétale, iRIG, CEA-Grenoble, Grenoble, France.
- 8Institut de Génétique Moléculaire de Montpellier, Univ. Montpellier, CNRS, Montpellier, France. edouard.bertrand@igh.cnrs.fr.
- 9Institut de Génétique Humaine, Univ. Montpellier, CNRS, Montpellier, France. edouard.bertrand@igh.cnrs.fr.
- 10Equipe labélisée Ligue Nationale Contre le Cancer, Montpellier, France. edouard.bertrand@igh.cnrs.fr.
- 11Aix Marseille Univ, CEA, CNRS, BIAM, UMR7265, SAVE (Signalisation pour l’Adaptation des Végétaux à leur Environnement), Saint-Paul lez Durance, France. lnussaume@cea.fr.